Nature Biotech:新工具可预测基因编辑的成功率
- 2023-06-20 00:00
- 来源:医药资讯网
- 阅读:300
在这项研究中,研究人员试图系统评估插入序列的长度和组成、细胞系、目标位点以及prime editor的不同版本如何影响插入效率。
为此,他们共设计了3,604条pegRNA,编码切口位点上游的插入物,这些插入物的长度从1nt到69nt不等,且GC含量不同。他们将序列插入两个细胞系(HEK293T和HAP1细胞)中,靶向四个目标位点(HEK3、EMX1、FANCF和CLYBL)。一周后,他们对这些细胞进行基因组测序,观察编辑是否成功。评估插入效率的整体策略详见图1。
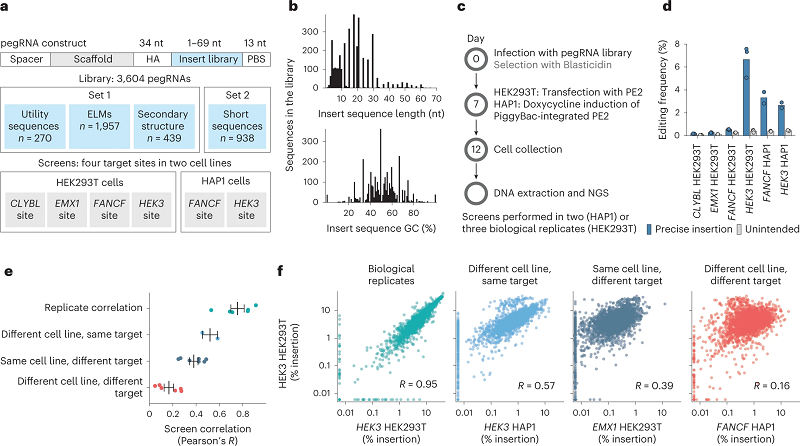
图1 prime插入效率的高通量测定
由于插入率的变化跨越了三个数量级,于是研究人员试图了解相关的特征,首先是插入物的长度。他们在HEK293T细胞中发现了两个特点:3和4nt序列的插入率高于其他序列;15-21nt序列的插入率高于周围的序列。不过在HAP1细胞中,1-4 nt短序列的插入率并没有高于较长序列。他们将其归因于错配修复(MMR)系统,因为HEK293T细胞在MMR上存在部分缺陷。敲除错配修复基因MLH1的HAP1细胞也证明了这一点,表明MMR系统阻碍了短序列的插入。
之后,他们分析了prime editing的不同步骤如何影响序列的插入率。他们发现,若pegRNA中带有四个或更多的连续腺嘌呤,则插入率会大大下降。此外,prime editing中的另一个重要步骤是带有5 flap(包含野生型序列)和3 flap(包含插入物)的中间产物之间达到平衡,而5 flap核酸酶FEN1及3 flap核酸酶TREX1和TREX2介导了这种平衡。他们发现,3 flap核酸酶TREX1和TREX2抑制了较长序列的插入。
同时,插入序列的核苷酸组成和二级结构也会影响插入率。研究人员发现,prime editing系统对胞嘧啶有着明显的偏好。插入序列中每增加1%的胞嘧啶,则插入率平均增加2.2%。相反,腺嘌呤和胸腺嘧啶的百分比则降低了各个位点的插入率。此外,他们还发现,结构强度更高的序列能够被更有效地插入。
在此过程中,他们使用了Twist Bioscience提供的寡核苷酸池。据Twist介绍,他们独特的硅基DNA合成平台能够在单次运行中生成超过一百万条寡核苷酸,对数量几乎没有限制,而且寡核苷酸池均一,让人们对实验结果充满信心。此外,实验中使用的多个载体和基因片段也是由Twist Bioscience提供的。
