微生物学领域数据库和分析系统建设的研究进展
- 2022-04-11 00:00
- 来源:医药资讯网
- 阅读:497
中国科学院计算机网络信息中心大数据技术与应用开发部、微生物研究所等。在微生物学领域的数据库和分析系统建设方面取得了新的进展,提出了利用语义网技术构建知识图谱的方法,可以将冠状病毒相关的毒株、基因组、蛋白质序列、蛋白质结构、抗体、文献、专利等多源异构数据映射到资源描述框架(RDF),构建基于语义网框架的gcCov知识图谱数据库。GcCov包含超过6000万个语义三元组。通过多源异构数据的语义集成,GCCOV支持大规模数据驱动的知识发现。具有分析基因、结构、抗体等数据相关性的能力,有助于推动未来病毒基础机制研究和药物、疫苗设计。相关研究成果发表在mLife上。
近几十年来,冠状病毒持续威胁全球公共健康。对新型冠状病毒的研究非常广泛,相关出版物的数量也迅速增加。海量的科研数据推动不同类型的研究整合成一个可搜索的语义互联数据集,这是相当具有挑战性的。目前可用的冠状病毒数据库主要集中在基因组分析领域(如CovDB1和ViPR2)或发表领域(如LitCovid3)。然而,这些数据库没有建立基因组数据与其他类型信息(如论文、专利和抗体)之间的关联,这阻碍了进一步的知识发现。
语义网可以将分布式网络资源整合到共享本体的知识库中,研究对象之间的潜在关系是生物医学数据集成的有效解决方案。为了分析海量数据之间的关系,提出了一套管道方法,将不同来源的数据整合到语义web框架中。在此基础上,本研究构建了gcCov数据库,并利用LinkOpen数据提供了冠状病毒的广泛信息和关系。GcCov是首个也是唯一一个利用关联开放数据基于语义web框架发布的冠状病毒数据库,帮助科学家检测关联数据之间的联系,发现隐藏在海量数据中的新知识。GcCov为当前和治疗策略提供线索,是满足冠状病毒研究日益增长的信息需求的重要工具。
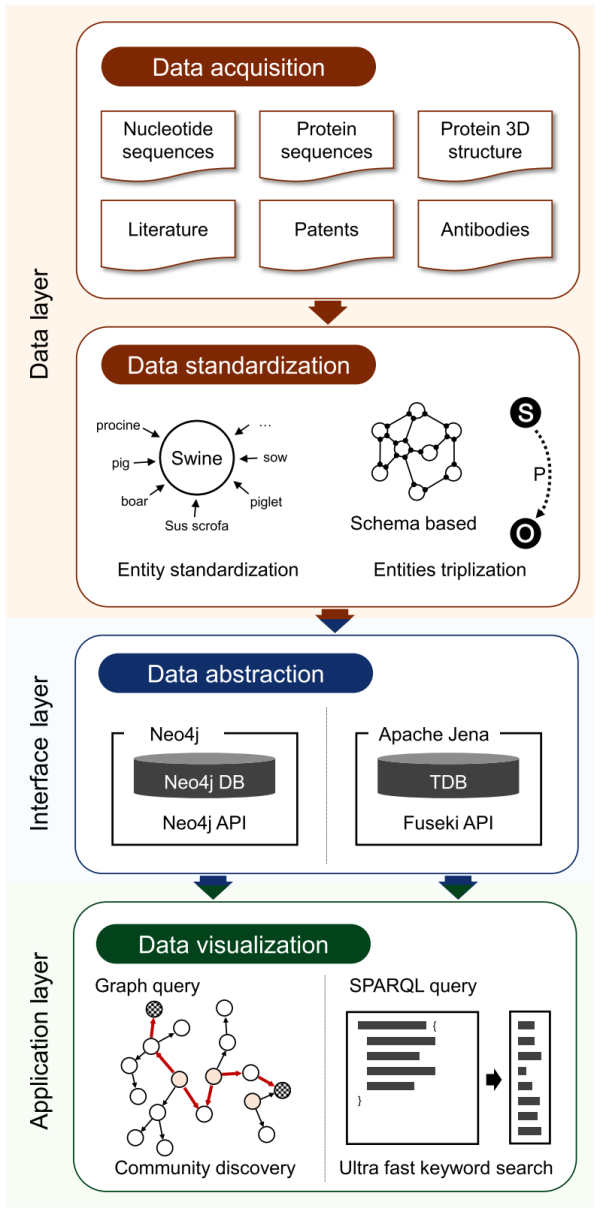
数据处理管道示意图
